
文章作者:王纯,张小雪,毛浩雨,咸逸,饶毅
点击下载原文:Development of a Genetically Encoded Sensor for Arginine
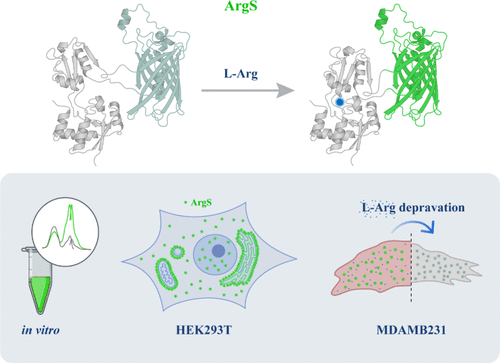
摘要: L-精氨酸(Arg)在多种代谢和生理过程中发挥重要作用,其浓度的变化与病理过程密切相关。尽管直接且实时测量生物系统中的Arg水平非常重要,但现有的Arg探针对L-鸟氨酸或L-赖氨酸也有响应。本文报道了一种新的Arg探针ArgS1。该探针对Arg表现出浓度依赖性的Ex488/405比率增加,其表观亲和力约为64 μM,动态范围(ΔR/R0)为3。ArgS1在细胞质和亚细胞器中对Arg均有响应。ArgS1成功监测了MDA-MB-231细胞(一种缺乏Arg合成关键酶——精氨琥珀酸合成酶1(ASS1)的乳腺癌细胞系,且适用于Arg耗竭疗法)中的Arg水平。研究发现,当细胞外Arg被耗竭后,MDA-MB-231细胞中的Arg水平下降,同时细胞活力也随之降低。当细胞中过表达ASS1时,Arg水平上升,细胞活力也得到增强。因此,ArgS1是一种在生理和病理相关动态范围内实时监测人类细胞中Arg水平的有效工具。
L-精氨酸(Arg)是一种条件性必需氨基酸,在人体中具有重要的生理作用[1, 2]。Arg的主要来源包括蛋白质降解、膳食摄入和从头合成[3]。Arg的合成涉及两种关键酶:精氨琥珀酸合成酶(ASS),它将L-瓜氨酸(Cit)转化为精氨琥珀酸;以及精氨琥珀酸裂解酶(ASL),它将精氨琥珀酸裂解为Arg[4, 5]。Arg在一氧化氮合酶(NOS)的催化下生成Cit和一氧化氮(NO),Arg是NO的唯一直接前体[6-9]。Arg还参与尿素循环,在精氨酸酶(ARGs)的作用下被水解为尿素和L-鸟氨酸(Orn)[10, 11],这对于氨的解毒至关重要[12]。精氨酸:甘氨酸脒基转移酶(AGAT)是一种线粒体酶,它催化Arg生成胍基乙酸,这是肌酸合成的直接前体[13-15]。Arg代谢紊乱与多种疾病相关[5, 16]。精氨酸酶1(ARG1)缺乏会导致高精氨酸血症,这是一种以进行性神经系统症状为特征的罕见遗传性代谢疾病[17]。Arg还与多种疾病的治疗相关,例如勃起功能障碍[18, 19]、高血压[20]和心力衰竭[21]。
癌细胞的生存和生长需要氨基酸[22-24],其中Arg是肿瘤微环境中的重要组成部分[25-27]。某些类型的癌症,如乳腺癌[28]、黑色素瘤[29]、肝细胞癌[30]、急性淋巴细胞白血病(ALL)[31]和急性髓系白血病(AML)[32],其ASS表达缺失。这些类型的癌细胞依赖摄取细胞外Arg以维持生存,因此限制Arg供应已被探索为癌症治疗中的一种潜在辅助治疗策略[27, 33]。在培养基中去除Arg或添加ADI-PEG20(聚乙二醇化精氨酸脱亚氨酶)可导致ASS1缺陷型癌细胞在体外被清除[28, 34]。系统性给予ADI或精氨酸酶以限制Arg可用性,已在肿瘤治疗的II期临床试验中进行了研究[35]。这些研究表明,ASS1缺陷是AML患者对ADI-PEG20单药治疗产生反应的必要条件,但并非充分条件。Arg的测量是确定患者是否适合接受ADI-PEG20治疗AML的重要指标。
传统的Arg测量方法包括样品的微透析[36, 37]和化学分析[38],这些方法无法研究细胞内分布的变化,也无法以必要的空间和时间分辨率分析Arg。荧光探针提供了非侵入性实时测量浓度及其时空变化的可能性。现有的基因编码Arg荧光探针基于Förster共振能量转移(FRET),其动态范围相对较小[39-41]。这些探针在体外报道的最大动态范围仅为约0.6[40]。更大的问题在于其特异性:这些探针不仅对Arg有响应,还对其他氨基酸(如Orn、L-赖氨酸(Lys)、L-谷氨酰胺(Gln)和L-组氨酸(His))有响应[39-41]。此外,这些探针对Arg的解离常数(Kd)(9.4或14 μM)[39, 40]不适合检测生理水平的Arg,因为血浆[42]和细胞质[43]中的Arg浓度通常约为100 µM。
因此,需要开发具有大动态范围和合适Kd的基因编码Arg特异性荧光探针,以用于生理和病理条件下Arg的检测。
结果
Arg探针的设计、优化及体外表征
我们基于大肠杆菌(E. coli)的artJ(一种周质结合蛋白,PBP)设计了一种基因编码的荧光Arg探针。artJ在结合Arg时会发生构象变化[44-46]。与其他探针的设计类似[47-50],我们将来自GRABDA2m的环状置换绿色荧光蛋白(cpEGFP)[51]整合到artJ中(图1A、B)。根据artJ和argT(一种Arg、Lys和Orn结合蛋白)的晶体结构,其叶II的环区和铰链区在底物结合时会发生显著构象变化[45, 46, 52-56],这为cpEGFP的插入提供了潜在的位点。首先,我们在纯化蛋白上筛选了cpEGFP的插入位点(图1C)。artJ与cpEGFP之间的连接肽设计为柔性(Gly-Gly)或刚性(Pro-Pro)。基于初步筛选结果,我们选择了动态范围大于0.1的探针(图1D)。考虑到基于蛋白纯化的筛选方法耗时较长,且在体外响应的探针在细胞环境中可能表现不同,我们转而使用HEK293T细胞进行筛选。通过将探针与iSeroSnFR的膜靶向序列融合[57],我们将探针靶向到人胚胎肾293T(HEK293T)细胞的细胞膜上,以优化连接肽的长度和氨基酸组成,从而最小化细胞内Arg对探针响应的影响。我们发现,在HEK293T细胞膜上表现出良好响应的探针的响应值分别为0.4和0.6,分别命名为ArgS0.1和ArgS0.2(图1C、D,图S1C)。随后,我们优化了连接肽邻近位点,发现响应最佳的探针的响应值为0.9,命名为ArgS1(图1D,图S1C)。总结而言,通过对1200多种不同变体的筛选,我们最终确定了三种探针,分别命名为ArgS0.1、ArgS0.2和ArgS1(图1D)。
我们将这三种探针分别在大肠杆菌中表达并纯化,随后测试了它们对各种氨基酸的特异性。结果显示,所有三种ArgS探针均特异性地响应L-Arg,而对其他氨基酸的L型或D型均无响应(图1E,图S2A、B)。这三种ArgS探针均表现出比率特性,具有两个激发峰(分别位于400纳米(nm)和500 nm附近)和一个发射峰(位于515 nm附近)。当与1 mM Arg结合时,三种探针的400 nm激发峰降低,而500 nm激发峰增加(图1F,图S2C、D)。我们还测量了ArgS0.1、ArgS0.2和ArgS1的Arg结合亲和力,其Kd值分别为73 μM、367 μM和1141 μM,荧光峰值变化(ΔR/R0)分别为1.9、4.6和19(图1G,图S2E、F)。同时,我们评估了ArgS探针对精氨酸相关代谢物的特异性。ArgS0.1对胍丁胺、Cit和精氨琥珀酸表现出可检测的响应,而ArgS0.2和ArgS1仅对Cit和精氨琥珀酸有响应(图S3A、D、G)。在瓜氨酸(Cit)结合实验中,ArgS0.1、ArgS0.2和ArgS1的Kd分别为312 μM、1162 μM和2912 μM,其荧光峰值变化约为Arg的一半(图S3B、E、H)。对于精氨琥珀酸的结合,所有探针的Kd值均超过300 μM(图S3C、F、G)。为了减少Arg相关代谢物的潜在干扰,我们通过LC-MS定量分析了HEK293T和MDA-MB-231细胞中这些代谢物的细胞内浓度。在两种细胞系中,Cit和精氨琥珀酸的浓度均低于5 μM(HEK293T细胞中Cit为1.290 μM,MDA-MB-231细胞中Cit为3.920 μM;HEK293T细胞中精氨琥珀酸为1.556 μM,MDA-MB-231细胞中精氨琥珀酸为4.156 μM),均低于ArgS探针的检测阈值(图S3J)。因此,在检测HEK293T或MDA-MB-231细胞中的Arg水平时,Cit或精氨琥珀酸的潜在干扰可忽略不计。此外,基于嗜热脂肪土芽孢杆菌(Geobacillus stearothermophilus)artJ的晶体结构[45],我们开发了三种ArgS1探针的突变版本。其中,双点突变版本ArgS1-F51L E114L对Arg几乎无响应(图S4C)。因此,该版本探针ArgS1-F51L E114L被命名为ArgS1-C,并作为后续实验的对照。为了研究pH依赖性,我们在pH 4至9的范围内测试了ArgS1和ArgS1-C。ArgS1在pH 7至8之间对Arg的响应最佳(图S4A),而ArgS1和ArgS1-C的apo形式在整个测试范围内表现出相似的pH依赖性行为(图S4B)。
Arg探针在HEK293T细胞质及亚细胞器中检测Arg变化
为了研究Arg探针是否能够在哺乳动物细胞中检测Arg,我们在HEK293T细胞的细胞质中表达了ArgS0.1、ArgS0.2和ArgS1(图2A)。Arg转运蛋白SLC7A1[58]和SLC7A2[59]在HEK293T细胞中高表达(图S5A)。当细胞外Arg浓度升高至1 mM时,我们立即观察到ArgS1、ArgS0.1和ArgS0.2在405 nm处的荧光强度下降,而在488 nm处的荧光强度增加(图2B,图S6A)。这表明所有三种探针(ArgS1、ArgS0.1和ArgS0.2)均对Arg的增加作出了响应,其最大动态范围分别为3.3、0.3和1.1(图2D,图S6D、E)。当Arg探针达到其峰值动态范围后,我们将灌注液切换回磷酸盐缓冲液(PBS),观察到三种探针在405 nm处的荧光强度立即增加,而在488 nm处的荧光强度下降,其响应恢复到基线值(图2C,图S6B、C)。
为了在HEK293T细胞质中进行原位滴定以测量ArgS1的Arg结合亲和力,我们用毛地黄皂苷(digitonin)透化细胞膜,并向细胞中添加不同浓度(1至∼60000 μM)的Arg(图S5B)。通过绘制表达ArgS1的细胞平均响应与Arg浓度的关系,获得了原位校准曲线。因此,我们测量了ArgS1在HEK293T细胞质中的Arg结合亲和力,其Kd值为64 μM(图2E)。此外,为了最小化非特异性荧光信号变化,我们在HEK293T细胞中表达了非结合对照探针ArgS1-C。如图S5D所示,ArgS1-C对1 mM Arg灌注无响应。
为了确定Arg探针是否能够检测由药物干预引起的HEK293T细胞中Arg的变化,我们在这些细胞中表达了ArgS1。我们利用精氨酸酶I和II抑制剂(Arginase Inhibitor 1, AI1)[60]和一氧化氮合酶(NOS)抑制剂L-NMMA[61-65]来调控Arg水平。如图2F所示,使用ArgS1检测到AI1或AI1与L-NMMA联合应用引起的细胞内Arg水平升高;然而,单独使用L-NMMA并未诱导类似的响应。这些结果表明,在HEK293T细胞中,精氨酸酶途径在Arg降解中的作用比NOS途径更为重要。这一结论与我们的转录组测序结果一致。在HEK293T细胞内,ARG2的表达水平最高,而ARG1和NOS2的表达水平非常低,NOS1则未表达(图S5C)。因此,ArgS1探针有效地监测了HEK293T细胞中药理干预引起的Arg浓度变化。
为了进一步研究Arg探针是否能够响应亚细胞器中的Arg,我们将ArgS1(图3)和ArgS0.1(图S7)靶向到不同的细胞器。我们将ArgS1和ArgS0.1分别与AKAP1的N端30个氨基酸引导序列[66]、p450的内质网(ER)靶向基序[67]、LAMP1衍生的序列[68]或核定位信号[69]融合,以定位到线粒体胞质侧(Mito-ArgS1和Mito-ArgS0.1)、ER胞质侧(ER-ArgS1和ER-ArgS0.1)、溶酶体(Lyso-ArgS1和Lyso-ArgS0.1)或细胞核(Nuc-ArgS1和Nuc-ArgS0.1)(图3A)。为了评估ArgS1在线粒体、ER、溶酶体和细胞核中的定位,我们分别使用Mito-Tracker、ER-Tracker、Lyso-Tracker和NucRed作为对照。结果显示,ArgS探针与相应的细胞器标记物高度共定位(图3B,图S7A)。当向培养HEK293T细胞的培养基中灌注1 mM Arg时,定位在细胞器中的ArgS探针也对Arg水平的变化作出了响应(图3C-F,图S7B-E)。Mito-ArgS1、ER-ArgS1、Lyso-ArgS1和Nuc-ArgS1探针的最大动态范围分别为3.7、3.8、3.2和3.8(图3G-J),而Mito-ArgS0.1、ER-ArgS0.1、Lyso-ArgS0.1和Nuc-ArgS0.1探针的最大动态范围分别为0.5、0.5、0.4和0.6(图S7F-I)。这些结果表明,ArgS探针在线粒体、ER和溶酶体附近区域以及细胞核内的响应相似。因此,我们成功开发了能够监测哺乳动物细胞质和亚细胞器中Arg的ArgS探针。
细胞外Arg剥夺导致癌细胞细胞内Arg水平下降
Arg饥饿正被探索为一种针对ASS1缺陷型癌症的潜在治疗策略[28, 70](图4A)。据报道,乳腺癌细胞系MDA-MB-231不表达ASS1[28](图4B)。我们首先验证了Arg剥夺对细胞活力的影响。Arg剥夺24小时后,MDA-MB-231细胞的活力下降(图4C),这与之前的报道一致[28]。此外,过表达ASS1能够回补MDA-MB-231细胞对Arg饥饿的敏感性(图4B、C)。为了观察细胞内Arg水平,我们开发了在MDA-MB-231细胞中稳定表达ArgS1的细胞系(图4A)。我们发现,Arg剥夺后,ArgS1探针的488/405比率及其衰减速率均显著下降,表明MDA-MB-231细胞内的Arg水平显著低于对照组(图4D、E,图S8B)。此外,Arg剥夺后,ASS1过表达组的488/405比率高于ASS1缺陷组,表明通过ASS1过表达回补了Arg水平(图4D、E)。此外,为了进一步减少非特异性荧光信号变化,我们在MDA-MB-231细胞中表达了ArgS1-C。如图S8A所示,ArgS1-C对1 mM Arg灌注也无响应。因此,ArgS1探针可用于监测癌细胞中的Arg水平。
讨论
为了将ArgS探针与其他Arg探针进行比较,我们整理了各种Arg探针的特性,如表S1所示。例如,基于glnH的QBP/Citrine/ECFP探针对Arg的Kd为2.1 mM,动态范围为0.3[39]。此外,基于argT的FLIP-cpargT194探针对Arg的Kd为48 μM,动态范围为0.5[40]。然而,这些探针并非对Arg完全特异:QBP/Citrine/ECFP还对Orn有响应,而FLIP-cpargT194对Orn和Lys均有响应[39, 40]。相比之下,FLIP-cpartJ185和FLIPR探针对Arg具有特异性,其Kd值分别为9.4 μM和14 μM,动态范围分别为0.5和0.3[40, 41]。由于这些探针对Arg的响应和特异性有限,它们并不适合检测哺乳动物细胞内Arg浓度的变化。相比之下,ArgS探针——ArgS0.1、ArgS0.2和ArgS1的Kd值分别为73 μM、367 μM和1141 μM,荧光峰值变化(ΔR/R0)分别为1.9、4.6和19。这些响应优于以往的Arg探针,且ArgS探针特异性地响应Arg,而不会与其他氨基酸发生交叉反应。最近,研究人员报道了一种名为STAR的基因编码探针,它特异性地响应Arg,并能够在体外和体内监测Arg动态[71]。与STAR相比,ArgS1的荧光响应略大(ΔR/R0:19 vs. ~16,体外)。然而,与ArgS1不同,STAR不具备比率特性,因此需要额外的对照来校正探针表达水平的变化。
为了增强ArgS探针的最大响应,我们筛选了不同长度和组成的连接肽。结果表明,N端连接肽为2或3个氨基酸、C端连接肽为2个氨基酸时,响应更大,且N端连接肽的首个氨基酸优选为Gly。此外,在筛选过程的第4步中,测试了N端连接肽邻近的氨基酸残基。例如,将该残基替换为Ile显著提高了响应,从而开发出了ArgS1。尽管我们筛选了1200多个候选变体,但通过进一步扩展筛选范围以包括更多邻近连接肽位点,仍有可能实现进一步改进。
具体而言,HEK293T细胞质中的ArgS1的Kd值为64 μM,高于体外测量的值。类似地,iGluSnFR探针在HEK293T细胞或培养的海马神经元中的亲和力也高于体外观察到的值[47, 49, 72]。这种差异可能是由于蛋白质在不同环境和检测系统中的暴露差异,也可能是细胞环境中的辅助因子增强了Arg结合的结果。然而,即使在体外实验中加入细胞裂解液后,测得的Kd仍处于毫摩尔范围,这表明需要进一步研究。因此,为了准确量化不同环境中的Arg水平,应在多种实验条件下测量Kd以考虑这些因素。此外,ArgS1对Cit和精氨琥珀酸表现出可检测的响应。在测量不同生物环境中的Arg水平时,必须确保Cit和精氨琥珀酸的浓度低于ArgS1探针的检测阈值。
关于ArgS探针在不同亚细胞位置的动态特性,Nuc-ArgS1和ER-ArgS1的Arg灌注时间比其他位置短,这可能反映了亚细胞间Arg代谢的差异(图3C-F)。此外,我们尝试确定每个亚细胞位置的基线Arg浓度;然而,我们不确定灌注前的初始比率是否准确反映了基线浓度,因为探针游离形式的荧光比率可能在不同亚细胞区室中有所不同。此外,在每个亚细胞位置除去Arg以获得ArgS探针的游离形式具有挑战性。因此,我们选择在获得更可靠的方法来获取这些区室中ArgS探针的游离形式之前,不对亚细胞位置的Arg浓度进行量化。
Arg在生理和病理过程中发挥着关键作用;然而,其许多功能仍未得到充分理解。例如,星形胶质细胞和神经元之间Arg转运的机制尚不清楚,其作为信号分子的作用也未明确定义。此外,Arg浓度的变化可以与其他分子(如Ca²⁺、cAMP和Glu)一起使用互补的荧光探针进行监测。我们希望ArgS探针能够有助于阐明Arg代谢的机制,并为其生理和病理作用提供更深入的见解。
结论
总之,ArgS1能够实时检测多种哺乳动物细胞中Arg水平的变化,为基础研究和临床应用提供了宝贵的工具。这是首个能够动态监测哺乳动物细胞内Arg浓度变化的基因编码荧光探针。此外,ArgS1在亚细胞器中对Arg表现出动态响应,并已成功用于监测MDA-MB-231乳腺癌细胞中的Arg水平。
致谢
我们感谢李毓龙教授提供GRABDA2m质粒。我们感谢国家蛋白质科学基础设施——北京基地北京大学分设施在Operetta高内涵成像实验提供的帮助。我们感谢北京脑科学与类脑研究所生物质谱中心于晓倩老师在LC-MS实验提供的帮助。
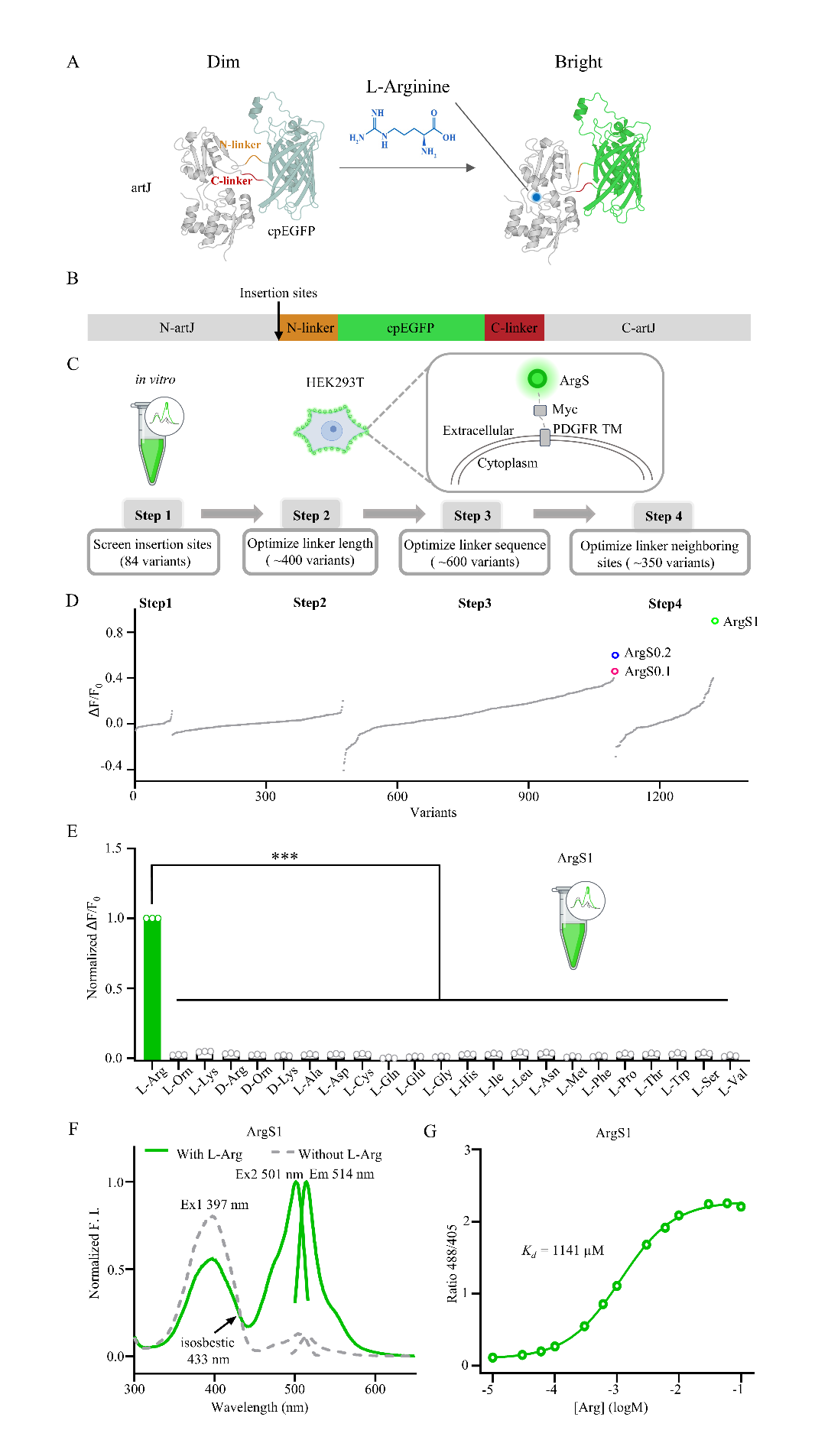
图1 ArgS探针的体外测量
A, B ArgS探针的设计。通过不同的插入位点将cpEGFP整合到大肠杆菌artJ蛋白中。我们将cpEGFP与artJ之间的氨基酸连接肽称为N端连接肽和C端连接肽。该模型使用AlphaFold2生成。
C ArgS探针的筛选流程。第1步是在纯化蛋白上进行插入位点筛选,第2至第4步是在HEK293T细胞膜上进行筛选,其中第2步和第3步分别优化连接肽的长度和组成,第4步优化连接肽邻近位点。
D ArgS探针的筛选结果。ArgS0.1、ArgS0.2和ArgS1探针分别用洋红色、蓝色和绿色突出显示。
E ArgS1探针的氨基酸特异性。对1 mM各种氨基酸的响应归一化为对1 mM L-Arg的响应。每组数据点n = 3;数据以均值±标准误(SEM)表示。
F ArgS1探针在有无1 mM Arg条件下的激发和发射光谱。
G ArgS1探针在不同浓度Arg暴露下的剂量响应曲线。每组数据点n = 6;数据以均值±标准误(SEM)表示。
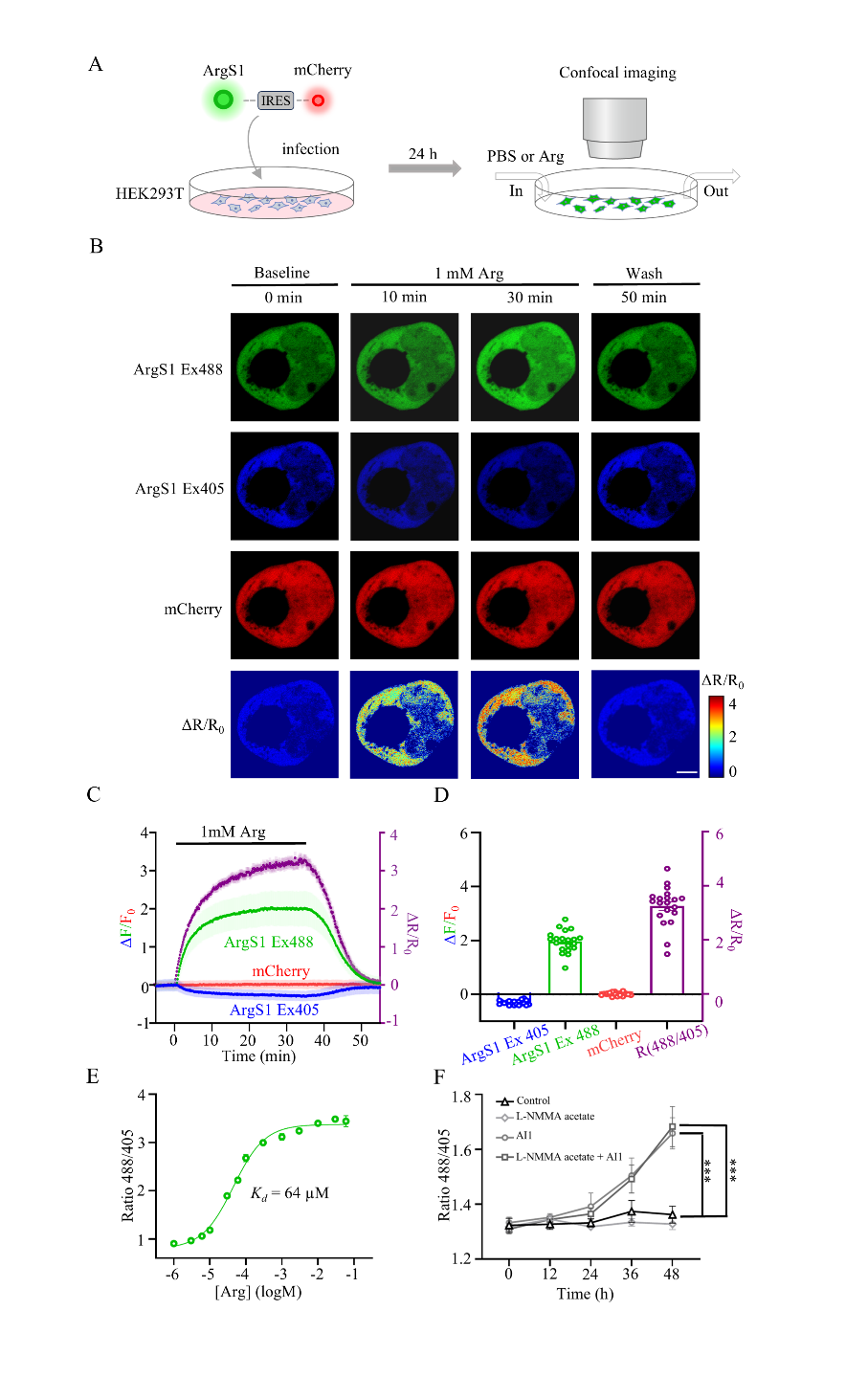
图2 ArgS1探针在HEK293T细胞质中对Arg浓度变化的响应
A ArgS探针在HEK293T细胞质中成像的示意图。在HEK293T细胞的细胞质中,C端融合IRES-mCherry序列的ArgS探针被转染,随后在不同溶剂灌注条件下使用共聚焦显微镜进行成像。
B 代表性图像显示ArgS1探针在HEK293T细胞质中对PBS或1 mM Arg变化在不同时间点(0分钟、10分钟、30分钟和50分钟)的响应。ArgS1探针的荧光在两个通道中可见:ArgS1 Ex488(激发波长488 nm)显示为绿色,而ArgS1 Ex405(激发波长405 nm)显示为蓝色。此外,通过IRES序列融合到ArgS探针C端的mCherry在mCherry通道中以红色显示。ArgS1探针的Ex488/Ex405比率变化进一步在ΔR/R0通道中可视化。比例尺:10 μm。
C ArgS1在PBS或1 mM Arg中荧光响应的平均轨迹。我们在0分钟时将1 mM Arg灌注到表达ArgS1探针的HEK293T细胞中,并在35分钟时切换为PBS缓冲液。ArgS1探针在405 nm激发波长下的ΔF/F0以蓝色表示,而在488 nm波长下的ΔF/F0以绿色表示。mCherry的ΔF/F0以红色表示。紫色表示ΔR/R0(右侧Y轴)。每组n = 20个细胞;数据以均值±标准误(SEM)表示。
D ArgS1探针对Arg灌注的最大响应。每组n = 20个细胞;数据以均值±标准误(SEM)表示。
E ArgS1探针在HEK293T细胞质中的剂量依赖曲线及相应的Kd值。每组n = 600个细胞(来自6个孔);数据以均值±标准误(SEM)表示。
F HEK293T细胞中Arg浓度的药理学改变。ArgS1探针的488/405比率在分别与500 μM抑制剂孵育12、24、36和48小时前后的变化。对照组以黑色等边三角形表示,单独L-NMMA组以灰色菱形表示,单独AI1组以灰色圆形表示,L-NMMA和AI1联合组以深灰色方形表示。每组n = 600个细胞(来自6个孔);数据以均值±标准误(SEM)表示。
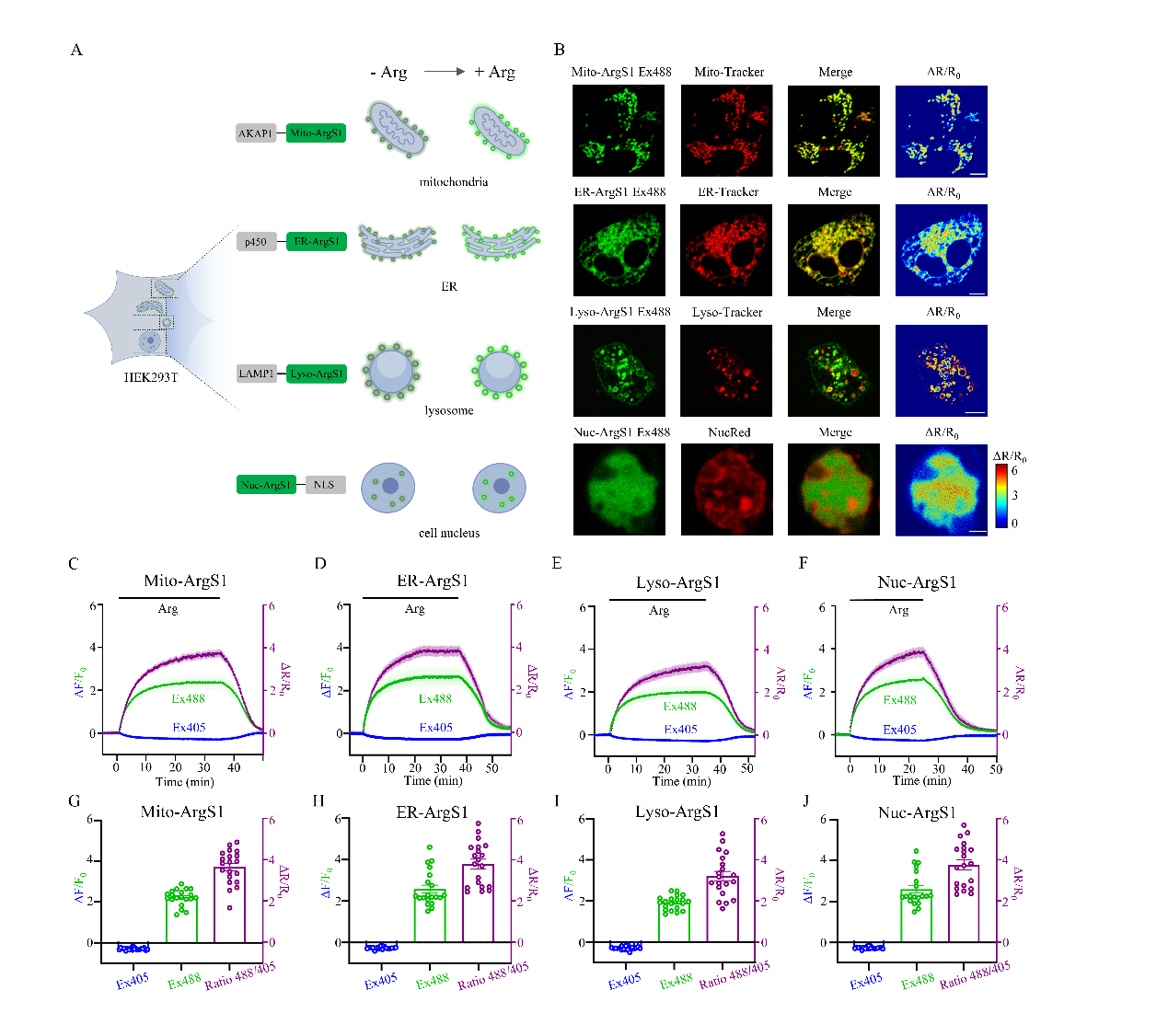
图3 ArgS1探针在HEK293T细胞器中对1 mM Arg灌注的响应
A ArgS1探针在不同细胞器中表达的示意图:线粒体、内质网(ER)、溶酶体和细胞核。
B ArgS1探针定位在不同细胞区室的图像:线粒体外膜的胞质侧(Mito-ArgS1)、ER膜的胞质侧(ER-ArgS1)、溶酶体膜的胞质侧(Lyso-ArgS1)以及细胞核内(Nuc-ArgS1)。ArgS1探针的荧光在ArgS1通道中以绿色表示。细胞器特异性标记物,包括线粒体的Mito-Tracker、ER的ER-Tracker、溶酶体的Lyso-Tracker和细胞核的NucRed,在细胞器标记通道中以红色表示。ArgS1探针荧光与细胞器标记荧光的合并图像显示在Merge通道中。ArgS1探针对Arg灌注的响应在ΔR/R0通道中表示。比例尺:5 μm。
C-F 定位在不同细胞区室的ArgS1在PBS或1 mM Arg中荧光响应的平均轨迹。我们在0分钟时将1 mM Arg灌注到表达Mito-ArgS1、ER-ArgS1、Lyso-ArgS1和Nuc-ArgS1探针的HEK293T细胞中,并在达到最大响应后分别切换为PBS缓冲液。ArgS1探针在405 nm激发波长下的ΔF/F0以蓝色表示,而在488 nm波长下的ΔF/F0以绿色表示。ΔR/R0以紫色表示。每组n = 20个细胞;数据以均值±标准误(SEM)表示。
G-J Mito-ArgS1、ER-ArgS1、Lyso-ArgS1和Nuc-ArgS1对1 mM Arg灌注的最大响应。每组n = 20个细胞;数据以均值±标准误(SEM)表示。
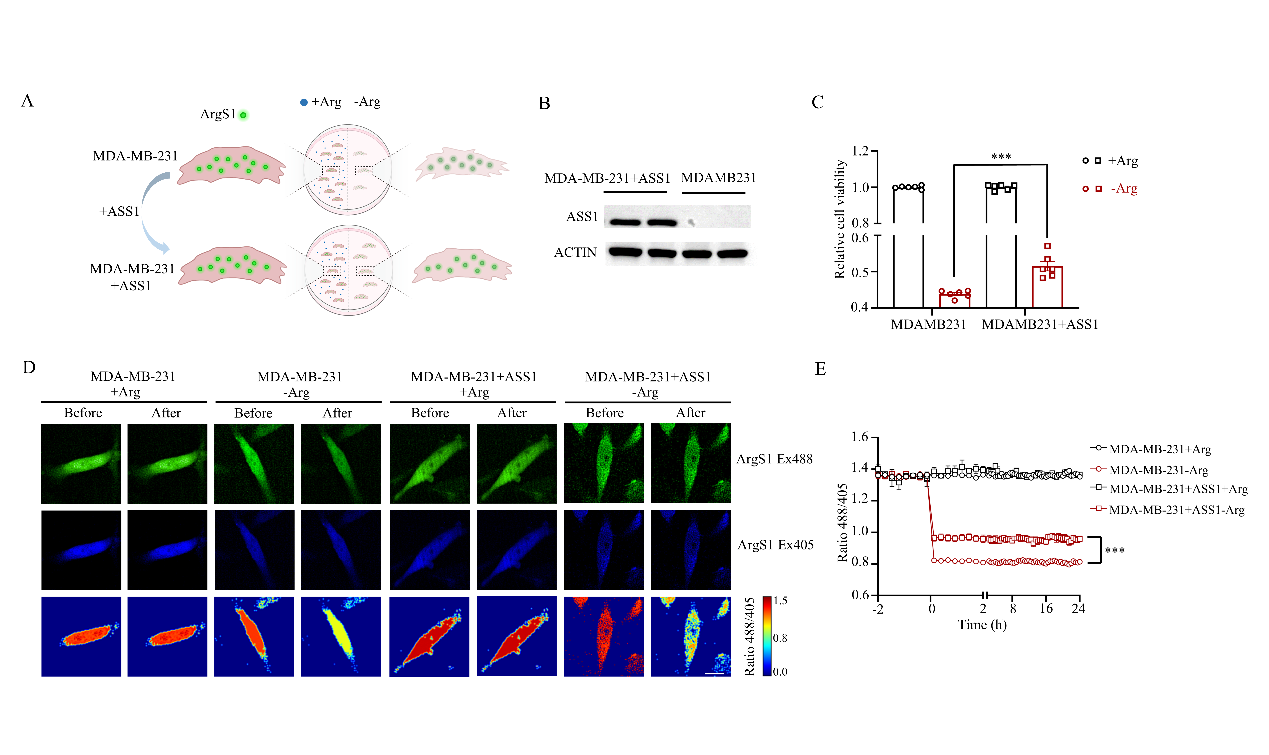
图4 MDA-MB-231细胞中Arg的成像
A ArgS1探针在MDA-MB-231细胞中表达的示意图,包括有和无Arg饥饿条件下的情况。
B MDA-MB-231细胞和ASS1过表达的MDA-MB-231细胞中ASS1的表达水平。
C MDA-MB-231细胞(圆形)和ASS1过表达细胞(方形)在Arg饥饿(深红色)或完全DMEM对照组(黑色)处理24小时后的细胞活力。Arg饥饿组的细胞活力均归一化为完全DMEM对照组。每组n = 600个细胞(来自6个孔);数据以均值±标准误(SEM)表示。
D ArgS1探针在MDA-MB-231细胞和ASS1过表达的MDA-MB-231细胞中的荧光强度和响应变化在Arg饥饿30分钟前后成像。比例尺:10 μm。
E ArgS1探针在MDA-MB-231细胞和ASS1过表达的MDA-MB-231细胞中的488/405比率变化在Arg饥饿2小时前和24小时后每15分钟成像一次。每组n = 600个细胞(来自6个孔);数据以均值±标准误(SEM)表示。
参考文献
(1) Albaugh, V. L.; Stewart, M. K.; Barbul, A. Chapter 27 - Cellu-lar and Physiological Effects of Arginine in Seniors. In Nutrition and Functional Foods for Healthy Aging, Watson, R. R. Ed.; Aca-demic Press, 2017; pp 317-336.
(2) Albanese, A. A. The Amino Acid Requirements of Man. In Advances in Protein Chemistry, Anson, M. L., Edsall, J. T. Eds.; Vol. 3; Academic Press, 1947; pp 227-268.
(3) Albaugh, V. L.; Barbul, A. Arginine. In Reference Module in Life Sciences, Elsevier, 2017.
(4) Jackson, M. J.; Beaudet, A. L.; O'Brien, W. E. Mammalian urea cycle enzymes. Annual review of genetics 1986, 20, 431-464. DOI: 10.1146/annurev.ge.20.120186.002243 From NLM.
(5) Wu, G.; Bazer, F. W.; Davis, T. A.; Kim, S. W.; Li, P.; Marc Rhoads, J.; Carey Satterfield, M.; Smith, S. B.; Spencer, T. E.; Yin, Y. Arginine metabolism and nutrition in growth, health and disease. Amino acids 2009, 37 (1), 153-168. DOI: 10.1007/s00726-008-0210-y From NLM.
(6) Marletta, M. A.; Yoon, P. S.; Iyengar, R.; Leaf, C. D.; Wishnok, J. S. Macrophage oxidation of L-arginine to nitrite and nitrate: nitric oxide is an intermediate. Biochemistry 1988, 27 (24), 8706-8711.
(7) Palmer, R. M. J.; Ashton, D. S.; Moncada, S. Vascular endo-thelial cells synthesize nitric oxide from L-arginine. Nature 1988, 333 (6174), 664-666.
(8) Bredt, D. S.; Snyder, S. H. Nitric oxide: a physiologic mes-senger molecule. Annual review of biochemistry 1994, 63 (1), 175-195.
(9) Moncada, S.; Higgs, E. A. Molecular mechanisms and thera-peutic strategies related to nitric oxide. The FASEB journal 1995, 9 (13), 1319-1330.
(10) Kossel, A.; Dakin, H. D. Über die Arginase. 1904.
(11) Krebs, H. A.; Henseleit, K. Hoppe-Seyler's Z. Physiol: 1932.
(12) Morris, S. M., Jr. Regulation of enzymes of the urea cycle and arginine metabolism. Annual review of nutrition 2002, 22, 87-105. DOI: 10.1146/annurev.nutr.22.110801.140547 From NLM.
(13) Wyss, M.; Kaddurah-Daouk, R. Creatine and Creatinine Metabolism. Physiological Reviews 2000, 80 (3), 1107-1213. DOI: 10.1152/physrev.2000.80.3.1107 (acccessed 2024/01/17).
(14) Foster, G. L.; Schoenheimer, R.; Rittenberg, D. Studies in protein metabolism: V. The utilization of ammonia for amino acid and creatine formation in animals. Journal of Biological Chemis-try 1939, 127 (1), 319-327.
(15) Rehberg, P. B. Studies on kidney function: the rate of fil-tration and reabsorption in the human kidney. Biochemical Jour-nal 1926, 20 (3), 447.
(16) Calabrò, R. S.; Gervasi, G.; Bramanti, P. L-Arginine and vascular diseases: lights and pitfalls! Acta bio-medica : Atenei Parmensis 2014, 85 (3), 222-228. From NLM.
(17) Schlune, A.; Vom Dahl, S.; Häussinger, D.; Ensenauer, R.; Mayatepek, E. Hyperargininemia due to arginase I deficiency: the original patients and their natural history, and a review of the literature. Amino acids 2015, 47 (9), 1751-1762. DOI: 10.1007/s00726-015-2032-z From NLM.
(18) Klotz, T.; Mathers, M. J.; Braun, M.; Bloch, W.; Engelmann, U. Effectiveness of Oral L-Arginine in First-Line Treatment of Erectile Dysfunction in a Controlled Crossover Study. Urologia Internationalis 2000, 63 (4), 220-223. DOI: 10.1159/000030454 (acccessed 1/19/2024).
(19) Kobori, Y.; Suzuki, K.; Iwahata, T.; Shin, T.; Sadaoka, Y.; Sato, R.; Nishio, K.; Yagi, H.; Arai, G.; Soh, S.; et al. Improvement of seminal quality and sexual function of men with oligoasthenoter-atozoospermia syndrome following supplementation with L-arginine and Pycnogenol®. Archivio Italiano di Urologia e Andro-logia 2015, 87 (3), 190-193. DOI: 10.4081/aiua.2015.3.190 (acccessed 2024/01/19).
(20) Gokce, N. L-Arginine and Hypertension. The Journal of Nutrition 2004, 134 (10), 2807S-2811S. DOI: https://doi.org/10.1093/jn/134.10.2807S.
(21) Drohomirecka, A.; Waś, J.; Wiligórska, N.; Rywik, T. M.; Komuda, K.; Sokołowska, D.; Lutyńska, A.; Zieliński, T. L-arginine and Its Derivatives Correlate with Exercise Capacity in Patients with Advanced Heart Failure. Biomolecules 2023, 13 (3). DOI: 10.3390/biom13030423 From NLM.
(22) Vettore, L.; Westbrook, R. L.; Tennant, D. A. New aspects of amino acid metabolism in cancer. British journal of cancer 2020, 122 (2), 150-156. DOI: 10.1038/s41416-019-0620-5 From NLM.
(23) Lieu, E. L.; Nguyen, T.; Rhyne, S.; Kim, J. Amino acids in cancer. Experimental & molecular medicine 2020, 52 (1), 15-30. DOI: 10.1038/s12276-020-0375-3 From NLM.
(24) Cormerais, Y.; Vucetic, M.; Pouysségur, J. Targeting amino acids transporters (SLCs) to starve cancer cells to death. Biochem-ical and biophysical research communications 2019, 520 (4), 691-693. DOI: 10.1016/j.bbrc.2019.10.173 From NLM.
(25) Ma, Q.; Wang, Z.; Zhang, M.; Hu, H.; Li, J.; Zhang, D.; Guo, K.; Sha, H. Targeting the L-arginine-nitric oxide pathway for can-cer treatment. Current pharmaceutical design 2010, 16 (4), 392-410. DOI: 10.2174/138161210790232121 From NLM.
(26) Lind, D. S. Arginine and cancer. J Nutr 2004, 134 (10 Suppl), 2837S-2841S; discussion 2853S. DOI: 10.1093/jn/134.10.2837S From NLM.
(27) Chen, C. L.; Hsu, S. C.; Ann, D. K.; Yen, Y.; Kung, H. J. Argi-nine Signaling and Cancer Metabolism. Cancers 2021, 13 (14). DOI: 10.3390/cancers13143541 From NLM.
(28) Qiu, F.; Chen, Y. R.; Liu, X.; Chu, C. Y.; Shen, L. J.; Xu, J.; Gaur, S.; Forman, H. J.; Zhang, H.; Zheng, S.; et al. Arginine starva-tion impairs mitochondrial respiratory function in ASS1-deficient breast cancer cells. Science signaling 2014, 7 (319), ra31. DOI: 10.1126/scisignal.2004761 From NLM.
(29) Chan, P. Y.; Phillips, M. M.; Ellis, S.; Johnston, A.; Feng, X.; Arora, A.; Hay, G.; Cohen, V. M. L.; Sagoo, M. S.; Bomalaski, J. S.; et al. A Phase 1 study of ADI-PEG20 (pegargiminase) combined with cisplatin and pemetrexed in ASS1-negative metastatic uveal mel-anoma. Pigment cell & melanoma research 2022, 35 (4), 461-470. DOI: 10.1111/pcmr.13042 From NLM.
(30) Ensor, C. M.; Holtsberg, F. W.; Bomalaski, J. S.; Clark, M. A. Pegylated arginine deiminase (ADI-SS PEG20,000 mw) inhibits human melanomas and hepatocellular carcinomas in vitro and in vivo. Cancer research 2002, 62 (19), 5443-5450. From NLM.
(31) De Santo, C.; Booth, S.; Vardon, A.; Cousins, A.; Tubb, V.; Perry, T.; Noyvert, B.; Beggs, A.; Ng, M.; Halsey, C.; et al. The argi-nine metabolome in acute lymphoblastic leukemia can be target-ed by the pegylated-recombinant arginase I BCT-100. Interna-tional journal of cancer 2018, 142 (7), 1490-1502. DOI: 10.1002/ijc.31170 From NLM.
(32) Mussai, F.; Egan, S.; Higginbotham-Jones, J.; Perry, T.; Beggs, A.; Odintsova, E.; Loke, J.; Pratt, G.; U, K. P.; Lo, A.; et al. Arginine dependence of acute myeloid leukemia blast prolifera-tion: a novel therapeutic target. Blood 2015, 125 (15), 2386-2396. DOI: 10.1182/blood-2014-09-600643 From NLM.
(33) Delage, B.; Fennell, D. A.; Nicholson, L.; McNeish, I.; Lem-oine, N. R.; Crook, T.; Szlosarek, P. W. Arginine deprivation and argininosuccinate synthetase expression in the treatment of can-cer. International journal of cancer 2010, 126 (12), 2762-2772. DOI: 10.1002/ijc.25202 From NLM.
(34) Cheng, C. T.; Qi, Y.; Wang, Y. C.; Chi, K. K.; Chung, Y.; Ouyang, C.; Chen, Y. R.; Oh, M. E.; Sheng, X.; Tang, Y.; et al. Argi-nine starvation kills tumor cells through aspartate exhaustion and mitochondrial dysfunction. Commun Biol 2018, 1, 178. DOI: 10.1038/s42003-018-0178-4 From NLM.
(35) Tsai, H. J.; Jiang, S. S.; Hung, W. C.; Borthakur, G.; Lin, S. F.; Pemmaraju, N.; Jabbour, E.; Bomalaski, J. S.; Chen, Y. P.; Hsiao, H. H.; et al. A Phase II Study of Arginine Deiminase (ADI-PEG20) in Relapsed/Refractory or Poor-Risk Acute Myeloid Leukemia Pa-tients. Scientific reports 2017, 7 (1), 11253. DOI: 10.1038/s41598-017-10542-4 From NLM.
(36) Siaghy, E. M.; Devaux, Y.; Schroeder, H.; Sfaksi, N.; Un-gureanu-Longrois, D.; Zannad, F.; Villemot, J. P.; Nabet, P.; Mertes, P. M. High-performance liquid chromatographic analysis of mus-cular interstitial arginine and norepinephrine kinetics. A micro-dialysis study in rats. Journal of chromatography. B, Biomedical sciences and applications 2000, 745 (2), 279-286. DOI: 10.1016/s0378-4347(00)00284-x From NLM.
(37) Watts, J.; Fowler, L.; Whitton, P. S.; Pearce, B. Release of arginine, glutamate and glutamine in the hippocampus of freely moving rats: Involvement of nitric oxide. Brain research bulletin 2005, 65 (6), 521-528. DOI: 10.1016/j.brainresbull.2005.03.011 From NLM.
(38) Seim, G. L.; Britt, E. C.; Fan, J. Analysis of Arginine Me-tabolism Using LC-MS and Isotopic Labeling. Methods in molecu-lar biology (Clifton, N.J.) 2019, 1978, 199-217. DOI: 10.1007/978-1-4939-9236-2_13 From NLM.
(39) Bogner, M.; Ludewig, U. Visualization of arginine influx in-to plant cells using a specific FRET-sensor. J Fluoresc 2007, 17 (4), 350-360. DOI: 10.1007/s10895-007-0192-2 From NLM.
(40) Okada, S.; Ota, K.; Ito, T. Circular permutation of ligand-binding module improves dynamic range of genetically encoded FRET-based nanosensor. Protein Sci 2009, 18 (12), 2518-2527. DOI: 10.1002/pro.266 From NLM.
(41) Whitfield, J. H.; Zhang, W. H.; Herde, M. K.; Clifton, B. E.; Radziejewski, J.; Janovjak, H.; Henneberger, C.; Jackson, C. J. Con-struction of a robust and sensitive arginine biosensor through ancestral protein reconstruction. Protein science : a publication of the Protein Society 2015, 24 (9), 1412-1422. DOI: 10.1002/pro.2721 From NLM.
(42) Lüneburg, N.; Xanthakis, V.; Schwedhelm, E.; Sullivan, L. M.; Maas, R.; Anderssohn, M.; Riederer, U.; Glazer, N. L.; Vasan, R. S.; Böger, R. H. Reference intervals for plasma L-arginine and the L-arginine:asymmetric dimethylarginine ratio in the Framingham Offspring Cohort. J Nutr 2011, 141 (12), 2186-2190. DOI: 10.3945/jn.111.148197 From NLM.
(43) Mao, Y.; Shi, D.; Li, G.; Jiang, P. Citrulline depletion by ASS1 is required for proinflammatory macrophage activation and immune responses. Mol Cell 2022, 82 (3), 527-541.e527. DOI: 10.1016/j.molcel.2021.12.006 From NLM.
(44) Rosen, B. P. Basic amino acid transport in Escherichia coli: properties of canavanine-resistant mutants. J Bacteriol 1973, 116 (2), 627-635. DOI: 10.1128/jb.116.2.627-635.1973 From NLM.
(45) Vahedi-Faridi, A.; Eckey, V.; Scheffel, F.; Alings, C.; Land-messer, H.; Schneider, E.; Saenger, W. Crystal structures and mu-tational analysis of the arginine-, lysine-, histidine-binding pro-tein ArtJ from Geobacillus stearothermophilus. Implications for interactions of ArtJ with its cognate ATP-binding cassette trans-porter, Art(MP)2. Journal of molecular biology 2008, 375 (2), 448-459. DOI: 10.1016/j.jmb.2007.10.049 From NLM.
(46) Soriani, M.; Petit, P.; Grifantini, R.; Petracca, R.; Gancitano, G.; Frigimelica, E.; Nardelli, F.; Garcia, C.; Spinelli, S.; Scarabelli, G.; et al. Exploiting antigenic diversity for vaccine design: the chla-mydia ArtJ paradigm. J Biol Chem 2010, 285 (39), 30126-30138. DOI: 10.1074/jbc.M110.118513 From NLM.
(47) Marvin, J. S.; Borghuis, B. G.; Tian, L.; Cichon, J.; Harnett, M. T.; Akerboom, J.; Gordus, A.; Renninger, S. L.; Chen, T. W.; Bargmann, C. I.; et al. An optimized fluorescent probe for visualiz-ing glutamate neurotransmission. Nat Methods 2013, 10 (2), 162-170. DOI: 10.1038/nmeth.2333 From NLM.
(48) Marvin, J. S.; Schreiter, E. R.; Echevarría, I. M.; Looger, L. L. A genetically encoded, high-signal-to-noise maltose sensor. Pro-teins 2011, 79 (11), 3025-3036. DOI: 10.1002/prot.23118 From NLM.
(49) Aggarwal, A.; Liu, R.; Chen, Y.; Ralowicz, A. J.; Bergerson, S. J.; Tomaska, F.; Mohar, B.; Hanson, T. L.; Hasseman, J. P.; Reep, D.; et al. Glutamate indicators with improved activation kinetics and localization for imaging synaptic transmission. Nat Methods 2023, 20 (6), 925-934. DOI: 10.1038/s41592-023-01863-6 From NLM.
(50) Marvin, J. S.; Shimoda, Y.; Magloire, V.; Leite, M.; Ka-washima, T.; Jensen, T. P.; Kolb, I.; Knott, E. L.; Novak, O.; Pod-gorski, K.; et al. A genetically encoded fluorescent sensor for in vivo imaging of GABA. Nat Methods 2019, 16 (8), 763-770. DOI: 10.1038/s41592-019-0471-2 From NLM.
(51) Sun, F.; Zhou, J.; Dai, B.; Qian, T.; Zeng, J.; Li, X.; Zhuo, Y.; Zhang, Y.; Wang, Y.; Qian, C.; et al. Next-generation GRAB sensors for monitoring dopaminergic activity in vivo. Nat Methods 2020, 17 (11), 1156-1166. DOI: 10.1038/s41592-020-00981-9 From NLM.
(52) Kang, C. H.; Kim, S. H.; Nikaido, K.; Gokcen, S.; Ames, G. F. Crystallization and preliminary X-ray studies of HisJ and LAO periplasmic proteins from Salmonella typhimurium. J Mol Biol 1989, 207 (3), 643-644. DOI: 10.1016/0022-2836(89)90475-0 From NLM.
(53) Kang, C. H.; Shin, W. C.; Yamagata, Y.; Gokcen, S.; Ames, G. F.; Kim, S. H. Crystal structure of the lysine-, arginine-, ornithine-binding protein (LAO) from Salmonella typhimurium at 2.7-A resolution. J Biol Chem 1991, 266 (35), 23893-23899. From NLM.
(54) Nikaido, K.; Ames, G. F. Purification and characterization of the periplasmic lysine-, arginine-, ornithine-binding protein (LAO) from Salmonella typhimurium. J Biol Chem 1992, 267 (29), 20706-20712. From NLM.
(55) Oh, B. H.; Pandit, J.; Kang, C. H.; Nikaido, K.; Gokcen, S.; Ames, G. F.; Kim, S. H. Three-dimensional structures of the periplasmic lysine/arginine/ornithine-binding protein with and without a ligand. J Biol Chem 1993, 268 (15), 11348-11355. From NLM.
(56) Oh, B. H.; Ames, G. F.; Kim, S. H. Structural basis for multi-ple ligand specificity of the periplasmic lysine-, arginine-, orni-thine-binding protein. J Biol Chem 1994, 269 (42), 26323-26330. From NLM.
(57) Unger, E. K.; Keller, J. P.; Altermatt, M.; Liang, R.; Matsui, A.; Dong, C.; Hon, O. J.; Yao, Z.; Sun, J.; Banala, S.; et al. Directed Evolution of a Selective and Sensitive Serotonin Sensor via Ma-chine Learning. Cell 2020, 183 (7), 1986-2002.e1926. DOI: 10.1016/j.cell.2020.11.040 From NLM.
(58) Afshinpour, M.; Mahdiuni, H. Arginine transportation mechanism through cationic amino acid transporter 1: insights from molecular dynamics studies. Journal of biomolecular struc-ture & dynamics 2023, 41 (23), 13580-13594. DOI: 10.1080/07391102.2023.2175374 From NLM.
(59) Banjarnahor, S.; Rodionov, R. N.; König, J.; Maas, R. Transport of L-Arginine Related Cardiovascular Risk Markers. Journal of clinical medicine 2020, 9 (12). DOI: 10.3390/jcm9123975 From NLM.
(60) Van Zandt, M. C.; Whitehouse, D. L.; Golebiowski, A.; Ji, M. K.; Zhang, M.; Beckett, R. P.; Jagdmann, G. E.; Ryder, T. R.; Sheeler, R.; Andreoli, M.; et al. Discovery of (R)-2-amino-6-borono-2-(2-(piperidin-1-yl)ethyl)hexanoic acid and congeners as highly po-tent inhibitors of human arginases I and II for treatment of myo-cardial reperfusion injury. J Med Chem 2013, 56 (6), 2568-2580. DOI: 10.1021/jm400014c From NLM.
(61) Cheng, J.; Valdivia, C. R.; Vaidyanathan, R.; Balijepalli, R. C.; Ackerman, M. J.; Makielski, J. C. Caveolin-3 suppresses late sodium current by inhibiting nNOS-dependent S-nitrosylation of SCN5A. J Mol Cell Cardiol 2013, 61, 102-110. DOI: 10.1016/j.yjmcc.2013.03.013 From NLM.
(62) Zhang, S.; Liu, Y.; Zhou, X.; Ou, M.; Xiao, G.; Li, F.; Wang, Z.; Wang, Z.; Liu, L.; Zhang, G. Sirtuin 7 Regulates Nitric Oxide Pro-duction and Apoptosis to Promote Mycobacterial Clearance in Macrophages. Front Immunol 2021, 12, 779235. DOI: 10.3389/fimmu.2021.779235 From NLM.
(63) Nakamura, T.; Prewitt, R. L. Effect of NG-monomethyl-L-arginine on arcade arterioles of rat spinotrapezius muscles. Am J Physiol 1991, 261 (1 Pt 2), H46-52. DOI: 10.1152/ajpheart.1991.261.1.H46 From NLM.
(64) Maggi, C. A.; Barbanti, G.; Turini, D.; Giuliani, S. Effect of NG-monomethyl L-arginine (L-NMMA) and NG-nitro L-arginine (L-NOARG) on non-adrenergic non-cholinergic relaxation in the circular muscle of the human ileum. Br J Pharmacol 1991, 103 (4), 1970-1972. DOI: 10.1111/j.1476-5381.1991.tb12361.x From NLM.
(65) Frey, C.; Narayanan, K.; McMillan, K.; Spack, L.; Gross, S. S.; Masters, B. S.; Griffith, O. W. L-thiocitrulline. A stereospecific, heme-binding inhibitor of nitric-oxide synthases. J Biol Chem 1994, 269 (42), 26083-26091. From NLM.
(66) Zhang, J. F.; Liu, B.; Hong, I.; Mo, A.; Roth, R. H.; Tenner, B.; Lin, W.; Zhang, J. Z.; Molina, R. S.; Drobizhev, M.; et al. An ultra-sensitive biosensor for high-resolution kinase activity imaging in awake mice. Nat Chem Biol 2021, 17 (1), 39-46. DOI: 10.1038/s41589-020-00660-y From NLM.
(67) Aryal, S. P.; Xia, M.; Adindu, E.; Davis, C.; Ortinski, P. I.; Richards, C. I. ER-GCaMP6f: An Endoplasmic Reticulum-Targeted Genetic Probe to Measure Calcium Activity in Astrocytic Process-es. Anal Chem 2022, 94 (4), 2099-2108. DOI: 10.1021/acs.analchem.1c04321 From NLM.
(68) Schmitt, D. L.; Curtis, S. D.; Lyons, A. C.; Zhang, J. F.; Chen, M.; He, C. Y.; Mehta, S.; Shaw, R. J.; Zhang, J. Spatial regulation of AMPK signaling revealed by a sensitive kinase activity reporter. Nature communications 2022, 13 (1), 3856. DOI: 10.1038/s41467-022-31190-x From NLM.
(69) Chen, M.; Sun, T.; Zhong, Y.; Zhou, X.; Zhang, J. A Highly Sensitive Fluorescent Akt Biosensor Reveals Lysosome-Selective Regulation of Lipid Second Messengers and Kinase Activity. ACS Central Science 2021, 7 (12), 2009-2020. DOI: 10.1021/acscentsci.1c00919.
(70) Cheng, C.-T.; Qi, Y.; Wang, Y.-C.; Chi, K. K.; Chung, Y.; Ouyang, C.; Chen, Y.-R.; Oh, M. E.; Sheng, X.; Tang, Y.; et al. Argi-nine starvation kills tumor cells through aspartate exhaustion and mitochondrial dysfunction. Communications Biology 2018, 1 (1), 178. DOI: 10.1038/s42003-018-0178-4.
(71) Li, R.; Li, Y.; Jiang, K.; Zhang, L.; Li, T.; Zhao, A.; Zhang, Z.; Xia, Y.; Ge, K.; Chen, Y.; et al. Lighting up arginine metabolism reveals its functional diversity in physiology and pathology. Cell Metabolism. DOI: 10.1016/j.cmet.2024.09.011.
(72) Marvin, J. S.; Scholl, B.; Wilson, D. E.; Podgorski, K.; Kazemipour, A.; Müller, J. A.; Schoch, S.; Quiroz, F. J. U.; Rebola, N.; Bao, H.; et al. Stability, affinity, and chromatic variants of the glutamate sensor iGluSnFR. Nat Methods 2018, 15 (11), 936-939. DOI: 10.1038/s41592-018-0171-3 From NLM.