
 zhangyu(at)cimrbj.ac.cn
zhangyu(at)cimrbj.ac.cn
张昱实验室开发并使用功能基因组筛选、多组学高通量测序技术和生物信息分析等方法,结合体内、体外小鼠模型和临床患者样本,研究肿瘤免疫和自身免疫疾病中关键分子的调控机制,研发肿瘤免疫治疗和自身免疫疾病治疗的新方法,并将研究成果进行临床转化。
研究方向包括肿瘤免疫和自身免疫疾病,主要包括肿瘤免疫中新机制和免疫检查点的鉴定及靶向方法;肿瘤免疫中B细胞的功能和调控;利用蛋白质工程、细胞工程和基因编辑治疗人类疾病。
1. 肿瘤免疫中新机制和免疫检查点的鉴定及靶向方法
近年来肿瘤的免疫治疗发展迅速,是目前治疗肿瘤最有效的策略之一。然而,这些免疫疗法只在某些肿瘤类型和某些患者中表现出显著疗效,其中的机制目前尚不清楚。本实验室通过将体外和体内的全基因组及定制的CRISPR/Cas9筛选相结合,并结合临床患者样本,旨在系统地鉴定出可调控肿瘤免疫治疗效果的新基因和相关通路。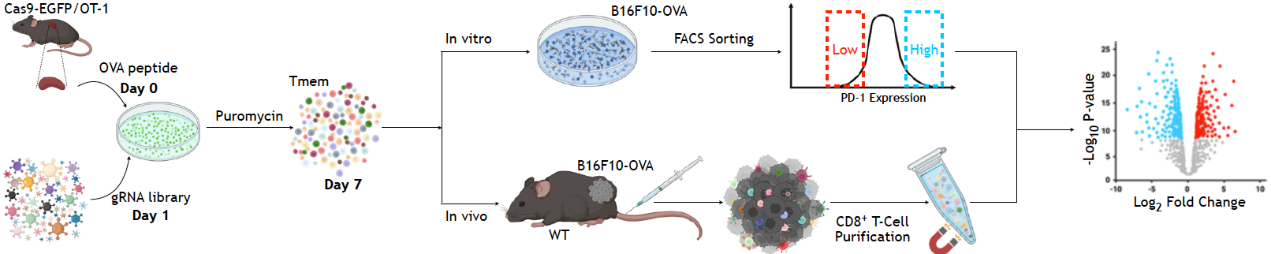

2. 肿瘤免疫中B细胞的功能和调控
众所周知,T淋巴细胞所介导的适应性细胞免疫在肿瘤免疫应答中具有重要作用,但B淋巴细胞在肿瘤的发生、发展及治疗中所起到的作用是近年来才逐渐被关注到的,且存在很多争议。近来的研究表明,在肿瘤患者接受免疫治疗时,肿瘤内的B细胞和三级淋巴结构(TLSs)可能与更好的预后相关,但其中的细胞和分子机制目前仍不清楚。本实验室正致力于将人类TLS的多组学分析与小鼠肿瘤模型相结合,以剖析B淋巴细胞在肿瘤免疫中的具体作用。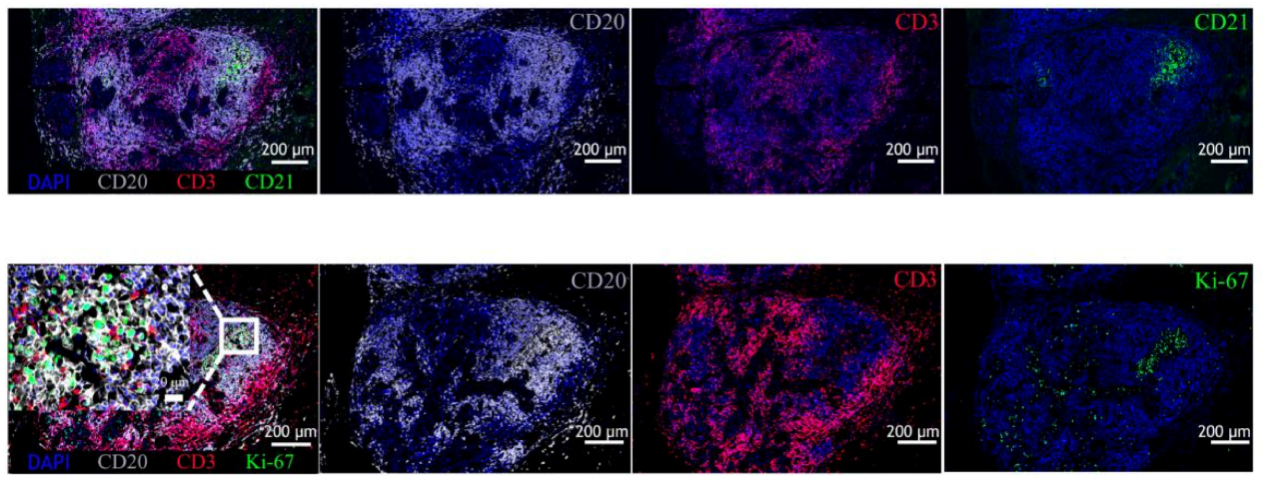

3. 利用蛋白质工程、细胞工程和基因编辑治疗人类疾病
随着对人类疾病中分子和细胞机制的深入了解,靶向特定致病细胞或其前体细胞的策略已逐渐发展为新一代的治疗手段。其中,蛋白质工程(如单克隆抗体、双特异性抗体、ADC等)及细胞疗法(如嵌合抗原受体T细胞(CAR-T)、CAR-NK、CAR-Macrophage等)对某些人类肿瘤展现出了惊人的疗效。近年来这类靶向致病细胞的疗法已扩展到其他非肿瘤疾病领域,如自身免疫病、心脏纤维化和衰老等相关疾病。本实验室正在开发新一代的治疗策略用于治疗人类疾病。

1. 组蛋白脱乙酰化酶6的功能(EMBO J,2003;G & D,2005;JBC,2006;MCB,2008)

2. 染色体转位的机制(Cell,2011;Cell,2012;Cell,2013;PNAS,2016)


4. 利用Cas9进行基因组标记(Genome Biology,2018a;Genome Biology,2018b;JMCB,2019)

5. 利用单细胞测序研究免疫细胞在疾病中的变化和功能(Circulation,2020; Nature Communications,2021)

Hu QT*, Hong Y*, Qi P*, Lu GQ, Mai XY, Xu S, He XY, Guo Y, Gao LL, Jing ZY, Wang JW, Cai T, Zhang Y#. Atlas of breast cancer infiltrated B-lymphocytes revealed by paired single-cell RNA-sequencing and antigen receptor profiling. Nature Communications, 2021, 12: 2186. DOI: 10.1038/s41467-021-22300-2
Hua XM*, Hu G*, Hu QT*, Chang Y, Hu YQ, Gao LL, Chen X, Yang PC, Zhang Y#, Li MY#, Song JP#. Single-Cell RNA Sequencing to Dissect the Immunological Network of Autoimmune Myocarditis. Circulation, 2020, 142: 384-400. DOI: 10.1161/CIRCULATIONAHA.119.043545
Han DQ, Hong Y, Mai XY, Hu QT, Lu GQ, Duan JZ, Xu JR, Si XF, Zhang Y#. Systematical study of the mechanistic factors regulating genome dynamics in vivo by CRISPRsie. Journal of Molecular Cell Biology, 2019, 11: 1018-1020. DOI: 10.1093/jmcb/mjz074
Duan JZ*, Lu GQ*, Hong Y*, Hu QT*, Mai XY, Guo J, Si XF, Wang FC, Zhang Y#. Live imaging and tracking of genome regions in CRISPR/dCas9 knock-in mice. Genome Biology, 2018, 19: 192. DOI: 10.1186/s13059-018-1530-1
Hong Y, Lu GQ, Duan JZ, Liu WJ, Zhang Y#. Comparison and optimization of CRISPR/dCas9/gRNA genome-labeling systems for live cell imaging. Genome Biology, 2018, 19: 39. DOI: 10.1186/s13059-018-1413-5
Lu GQ*, Duan JZ*, Shu S, Wang XX, Gao LL, Guo J, Zhang Y#. Ligase I and ligase III mediate the DNA double-strand break ligation in alternative end-joining. Proc Natl Acad Sci U S A, 2016, 113: 1256-1260. DOI: 10.1073/pnas.1521597113
Duan JZ*, Lu GQ*, Xie Z, Lou ML, Luo J, Guo L, Zhang Y#. Genome-wide identification of CRISPR/Cas9 off-targets in human genome. Cell Research, 2014, 24: 1009-1012. DOI: 10.1038/cr.2014.87
Zhang Y*, McCord RP*, Ho YJ, Lajoie BR, Hildebrand DG, Simon AC, Becker MS, Alt FW#, Dekker J#. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations. Cell, 2012, 148: 908-921. DOI: 10.1016/j.cell.2012.02.002
Chiarle R*, Zhang Y*#, Frock RL*, Lewis SM*, Molinie B, Ho YJ, Myers DR, Choi VW, Compagno M, Malkin DJ, Neuberg D, Monti S, Giallourakis CC#, Gostissa M#, Alt FW#. Genome-wide translocation sequencing reveals mechanisms of chromosome breaks and rearrangements in B cells. Cell, 2011, 147: 107-119. DOI: 10.1016/j.cell.2011.07.049