人类的组织修复和再生能力相当有限。相比之下,自然界中存在具有非凡再生能力的生物。其中,蝾螈类动物(如蝾螈和美西螈)被认为是再生能力最强的生物。它们能够在一生中重新长出各种极其复杂的结构,包括眼部组织、尾部、颌部、心脏大部分区域、部分神经系统以及完整的肢体。因此,蝾螈构成了一个理想的模型系统,用于研究如何实现身体结构的精确再生。
Yun实验室致力于利用可操作性强的蝾螈模型系统,旨在:揭示器官再生的细胞与分子机制;确定系统发育变化如何影响再生能力;探索再生与衰老之间的联系。
1. 再生过程中细胞可塑性的调控
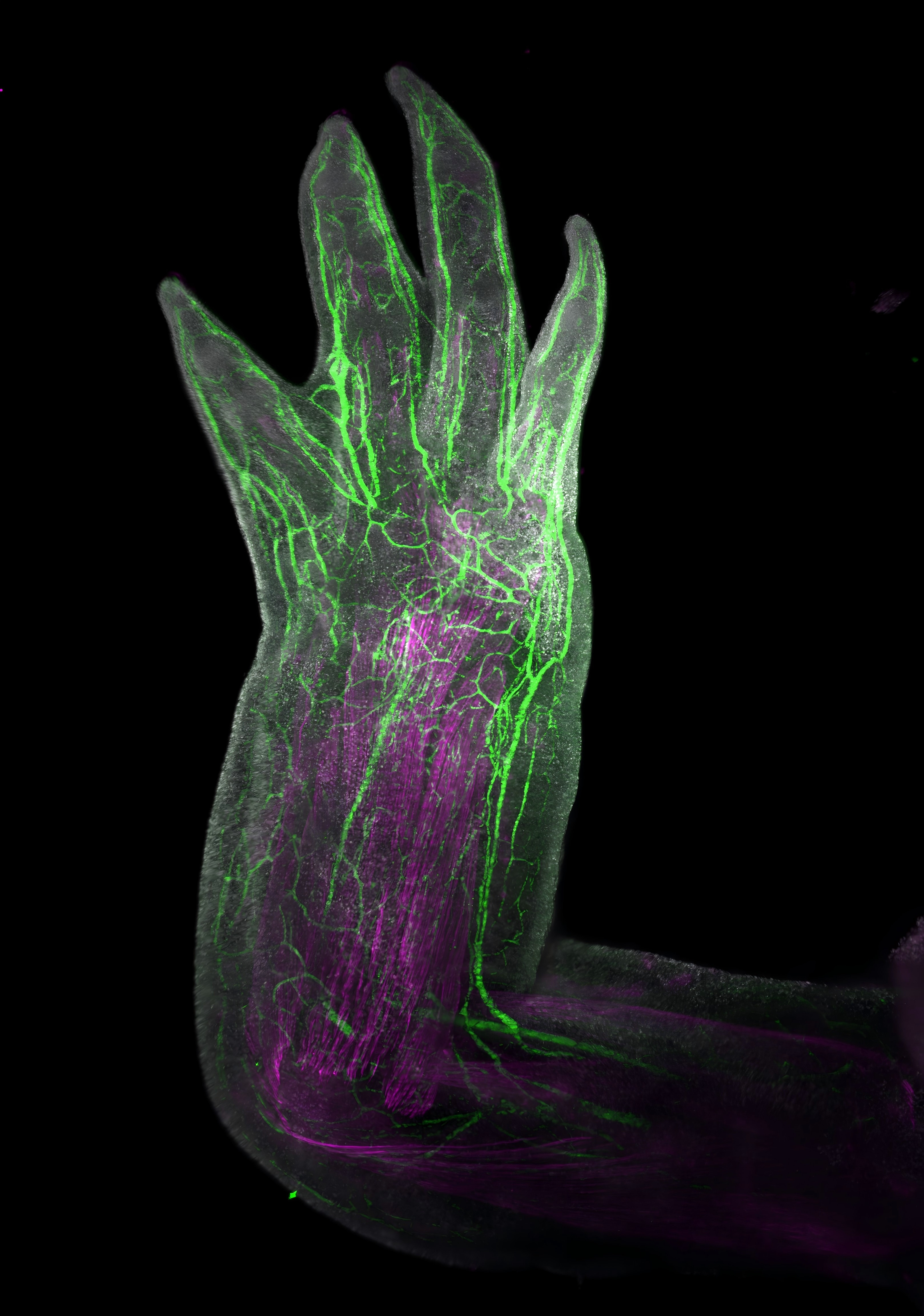

2. 新生胸腺再生的细胞与分子基础



3. 探究再生能力增强背景下的衰老机制
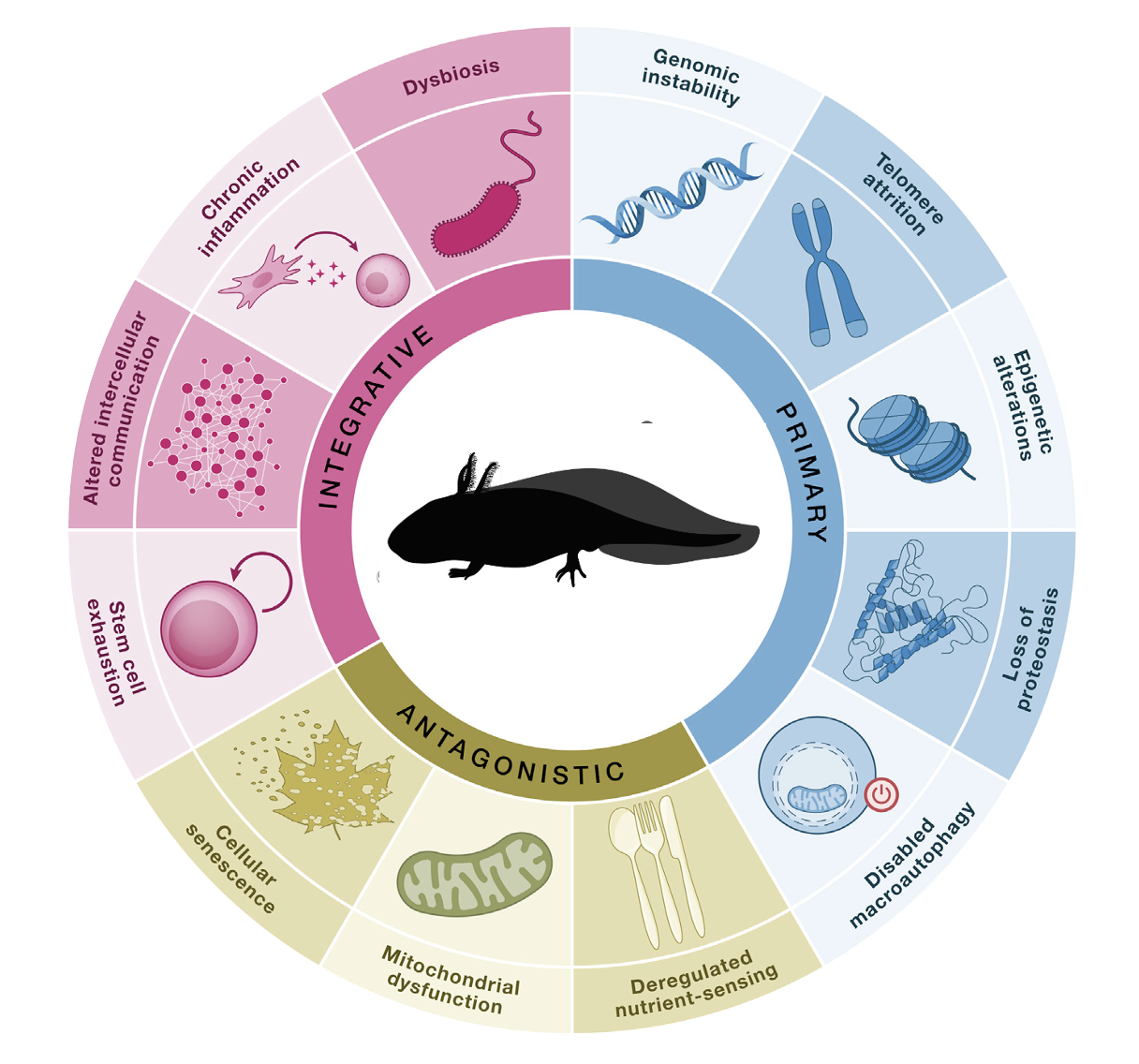

4. 无限再生能力的潜在机制



2. 发现了调控细胞去分化的内在与外在因子(PNAS, 2013; Stem Cell Reports, 2014; Aging Cell, 2023)及位置信息(Nature Commun., 2022)。

3. 揭示了衰老细胞在脊椎动物发育与再生中的关键作用(eLife, 2015; Development, 2017; Aging Cell, 2023; Developmental Cell, 2023),并为体内细胞衰老研究贡献了指导性规范(Cell, 2024)。

4. 揭示了双链断裂修复的基本机制,该机制对于维持基因组稳定性和避免衰老至关重要(Nature, 2009)。
